27.3.2020
Istället för att direkt döda skadliga bakterier med antibiotika kan man hitta sätt att blockera bakteriernas sätt att förvirra immunförsvaret och därmed låta immunförsvaret ta hand om bakterierna. Genom att försöka sätta sig in i processen att hitta lämpliga molekyler som kan designas till läkemedel kan man lära sig något om komplexiteten i forskningsområdet.
Antibiotikaresistenta bakterier kan utgöra ett globalt hot då inga kända läkemedel biter på dem. En orsak till deras uppkomst är överanvändning av antibiotika. Problemet uppstår eftersom antibiotika inte fungerar på precis alla bakterier, men på de bakterier den fungerar angriper den bakteriernas centrala livsfunktioner som DNA-syntesen, vilket helt enkelt innebär att de sårbara bakterierna dör ut medan de resistenta överlever och förökar sig.
– En version av detta är MRSA-bakterien, det vill säga meticillinresistenta Staphylococcus aureus, även kallad sjukhusbakterie. Meticillinresistens betyder att bakterien utvecklat resistens mot betalaktamantibiotika, alltså penicilliner, cefalosporiner och karbapenemer, säger Ida Alanko som är doktorand i farmaci vid Åbo Akademi.
Alanko arbetar tillsammans med sin professor i farmaci Outi Salo-Ahen på ett projekt som går ut på att hitta sätt att neutralisera MRSA-bakteriens sätt att angripa det mänskliga immunförsvaret.

Bakgrund: Staphylococcus aureus, eller gula stafylokocker, är en vanlig bakterie. Den uppskattas finnas på hälften av alla friska människor och lever på huden och i näsans slemhinnor – för det mesta i symbios och utan att skapa problem. Men den klassas också som patogen och kan latent förorsaka opportunistisk infektion i de flesta vävnader. MRSA, som däremot ofta skapar problem, är den antibiotikaresistenta stammen av gula stafylokocker.
Ett sätt att komma åt problemet med antibiotikaresistenta bakterier är att blockera bakteriernas möjligheter att sätta hinder i vägen för immunförsvarets vapen, utan att direkt döda bakterien.
Bakterierna sätter hinder för immunförsvaret genom att sprida så kallade virulensfaktorer. Virulensfaktorer är till exempel vissa typer av proteiner, toxiner och enzymer som hjälper bakterien att stanna kvar i värden och orsaka infektion utan att immunförsvarets tunga artilleri, de vita blodkropparna (leukocyter) kan äta upp dem.
Staphylococcus aureus kan uttrycka och utsöndra tiotals olika virulensfaktorer så som stafylokockala superantigenlika proteiner (SSL) – dessa proteiner hindrar alltså immunförsvaret från att komma åt bakterierna.
Blockera proteinbindning istället för att förinta bakterierna
Man känner till 14 olika typer av SSL-proteiner som Staphylococcus aureus utsöndrar. Dessa bakteriella molekyler binds till immunsystemets olika delar och stör därmed både människans medfödda och adaptiva immunförsvar. En möjlig uppgift för en forskare är då att försöka hitta sätt att neutralisera SSL-proteinerna. Hypotesen är att bakterien inte nödvändigtvis utvecklar resistens mot ett läkemedel som neutraliserar en virulensfaktor, detta eftersom bakterien inte är beroende av proteinets effekt för sin egen omedelbara överlevnad. Hypotesen säger vidare att ett SSL-blockerande läkemedel i kombination med antibiotika kunde vara en effektiv kur mot allvarliga infektioner förorsakade av Staphylococcus aureus.
Det protein Ida Alanko undersöker heter SSL5. SSL5, är en av de 14 SLL-proteinerna. SSL5 binds till och hämmar enzymet matrixmetalloproteinas 8 (MMP8). MMP8 är en viktig komponent i immunförsvarets aktivering av neutrofiler (det vill säga en av immunförsvarets vita blodkroppar) som angriper Staphylococcus aureus.
Eller för att vara exakt: Alanko undersöker bindningsmekanismen av virulensfaktorn SSL5 på enzymet MMP8.
Avsikten är att hitta exakt var på enzymet MMP8 virulensfaktorn ”dockar” – det vill säga hitta en strukturell region på enzymet MMP8 som har de egenskaper som krävs för att proteinet SSL5 ska fastna. Det är också viktigt att veta vilken region på SSL5 som binds till MMP8.
När man får reda på det kan man försöka hitta eller designa en annan molekyl som har samma ytegenskaper som bindningsregionen hos MMP8, och istället låta den binda till motsvarande bindningsregion på SSL5. Den molekylen kommer i så fall att blockera bindningen av SSL5 på enzymet MMP8. Då förblir enzymet MMP8 fritt att aktivera neutrofilerna till att angripa Staphylococcus aureus.
– Vad vi försöker ta reda på är alltså vilken typ av molekyl man kunde introducera för att ta enzymets plats så att SSL5 inte längre kan binda vid enzymet MMP8. Vi behöver alltså först hitta bindningsstället mellan SLL5 och MMP8 för att veta exakt hur bindningsytan ser ut just på det stället. Och när vi studerar ytan för det specifika stället där SSL5 binder vid MMP8 gör vi det för att kunna veta vilken typ av molekyl vi ska söka eller designa som ska ta MM8:s plats. Den molekylen ska alltså ha en passande struktur, en yta, som motsvarar bindningsstället vid SSL5.
För att få reda på en sådan sak behöver Alanko producera en tillräckligt stor mängd rena SSL5- och MMP8-proteiner, vilket är en lång process. När hon har tillräckligt mycket av båda proteinerna blandar hon dem så att de fäster vid varandra och sedan sänker hon ner proteinerna i en kristalliseringsvätska. Eller just den fasen sköts av en kristalliseringsrobot som finns i biokemisternas laboratorium i Biocity. Trots att Alanko behöver ”stora mängder” SLL5 och MMP8 är det ändå frågan om mikroskopiska doser.
Kristallisering och diffraktionsanalys
Kristalliseringsroboten doserar mikroskopiska mängder kristalliseringsmedel (saltlösning) i ett hundratal minimala behållare som innehåller proteinkomplexet SSL5-MMP8. Varje behållare har en egen komposition av kristalliseringsmedel.
– Man vet inte på förhand hur länge det tar för en specifik typ av saltlösningen tillsammans med proteinerna som ska undersökas att bilda en kristall. Det kan ta månader, ett år, eller två dagar. Vi måste testa olika typer av saltlösningar, eftersom man i det här fallet inte på förhand vet vilken komposition som fungerar bäst i kombination med proteinet man vill kristallisera, säger professor Outi Salo-Ahen.
Uppsättningen behållare förseglas och placeras sedan i ett så kallat kristalliseringshotell (ett slutet skåp) där varje enskild droppe som man väntar på att ska kristalliseras automatiskt fotograferas. Behållarna i hotellet kan övervakas online för att se när kristaller bildats.
– Man måste pröva sig fram. Inte ens då man har protokoll färdiga för kända proteiner kan man vara helt säker på vilken tid som krävs för kristalliseringsprocessen, eller om det fungerar över huvud taget.

När kristalliseringen skett beställer forskaren tid hos en synkrotron, exempelvis i Grenoble eller Lund, dit hon reser med kristallen eller kristallerna. Det protein som ska undersökas behöver ha kristalliserats för att kunna studeras i synkrotron.
I synkrotronen strålas kristallen. Utifrån hur strålningen diffrakterar, alltså hur strålningen sprids eller bryts, sluter sig mjukvara till var aminosyrorna finns och vad elektrondensiteten är. Detta jämförs sedan med ett (digitalt) bibliotek som i sin tur ger en tredimensionell struktur på aminosyrorna. Ifall man haft kristaller av ett protein-proteinkomplex eller protein-småmolekylkomplex så har man alltså fått en tredimensionell struktur av hela komplexet och då kan man direkt studera bindningsgränsytan mellan molekylerna.
– Det här kan ju låta enkelt, trots de många olika faserna. Men proteiner är oerhört komplexa och kan dessutom vara instabila – att man får entydiga och enkla data ur diffraktionsanalysen är inte självklart, säger Ida Alanko.
Proteiner är oerhört komplexa och kan dessutom vara instabila – att man får entydiga och enkla data ur diffraktionsanalysen är inte självklart
Om och när man har strukturen klar kan man skicka resultatet till biodatabasen Protein Data Bank (PDB) som finns på nätet. PDB ger verktyg att designa nya läkemedel av målproteinets med hjälp av målproteinets tredimensionella struktur eller hitta bindningsstället för ett tänkt läkemedel. PDB har en databas med experimentellt bestämda tredimensionella strukturer av biomakromolekyler (mestadels proteiner, också RNA och DNA). En stor del av strukturerna i PDB har man fått fram i röntgendiffraktionsanalyser av kristalliserade proteiner.
– Proteiner har en primär struktur som utgörs av ordningen på aminosyrorna den innehåller. Att bestämma strukturen innebär att modellera aminosyrorna i den tredimensionella rymden i denna ordning enligt den klargjorda elektrodensiteten. De sekundära proteinstrukturerna, de kallas alfahelixar och betaflak, knyts samman med vätebindningarna mellan aminosyrorna och den tertiära strukturen är den tredimensionella rymdstrukturen, säger Salo-Ahen.
– Man saknar ofta en experimentellt bestämd struktur av proteinkomplex, det vill säga protein bundna vid deras bindningspartner, man har bara individuella strukturer av obundna molekyler. Då kan man försöka hitta information om till exempel mutationsstudier som kan påvisa vilka aminosyror som är viktiga för bindningen av den andra molekylen. Från den tredimensionella strukturen av proteinet ser man var aminosyrorna ligger, vilket avslöjar den viktiga bindningsregionen. Om man inte har någon experimentell kunskap om aminosyrorna som deltar i bindningen mellan proteinerna kan man istället försöka räkna ut hur proteinytans elektrostatiska laddning ser ut hos båda bindningsparterna, och då, på basis av dessa laddningsvärden får man fram ytans form med datorprogram som kan förutsäga var dockningsprocessen, eller bindningsprocessen sker.
Om man inte ens har en tredimensionell struktur av målproteinet, i detta fall proteinerna SSL5 och MMP8, kan man använda proteiner som liknar dem som modell för att bygga tredimensionella modellstrukturer. När man har en experimentell struktur, eller en modell, av SSL5-MMP8-komplexet, kan man med datorhjälp söka småmolekyler (eller peptider) som kunde tänkas bindas till regionen där MMP8 binder vid SSL5. I en virtuell sållning dockar man en stor molekyldatabas till bindningsregionen av målmolekylen och får ett score, ett värde som för varje individuell databasmolekyl säger hur bra eller sannolik bindningen är.
När man hittat goda virtuella hits (träffmolekyler), alltså lovande molekylstrukturer med goda bindningsinteraktioner och högt ”dockningsscore”, kan man köpa eller syntetisera dessa molekyler i labbet och se om de fungerar. Men det finns miljoner olika alternativ att simulera, vilket kräver mycket datorkraft.
– För sådant jobb använder vi superdatorn vid CSC – IT Center for Science i Esbo. De har en ny maskin, ”Puhti”, som är till för att processera just sådana här stora datamängder, säger Salo-Ahen.
Även en annan väg
Just nu är Ida Alanko i färd med kristalliseringsprocessen. Men samtidigt undersöker hon ändra andra vägar för att blockera proteinet SSL5 från att påverka enzymet MMP8.
– Vi har ett samarbete med Åbo universitet som har ett antikroppsbibliotek. I antikroppsbiblioteket kan man försöka leta fram antikroppsfragment som kunde tänkas binda vid SSL5.
– Vi har hittat flera fragment som binder men vi vet inte ännu vilken funktion dessa fragment har, det vill säga kan de återhämta enzymet MMP8:s aktivitet?
Det man hoppas nå genom båda vägarna är att hitta en lämplig molekyl som binder på rätt plats. När man hittat rätt typ av molekyl gäller det att skaffa eller framställa den, testa om den binder till och hämmar funktionen hos SSL5 samt återhämtar aktiviteten hos MMP8, och sedan – vilket är en helt egen lång process – se vilka vägar man ska gå för att kunna framställa ett fungerande läkemedel.
Tillvägagångssätt 1
- Odla upp tillräcklig mängd rena MMP8 och SLL5 proteiner.
- Låt dem binda.
- Tillsätt kristalliseringsämne.
- Vänta på att proteinkomplexet kristalliseras i kristalliseringshotellet.
- När kristalliseringen skett: ta med kristallen till en synkrotron.
- Bestråla kristallen i synkrotronen.
- Analysera data från diffraktionen och räkna ut den tredimensionella strukturen.
- Använd den experimentella strukturen för att identifiera och studera bindningsregionen.
- Sålla molekyldatabaser virtuellt mot denna bindningsregion, kör databehandlingen i en superdator: du borde få ett antal kandidater som har ett bra bindningsvärde.
- Syntetisera eller köp vald molekyl.
- Gör labbexperiment.
- Om bra resultat: Voilà! Nu finns det en molekyl som är en god kandidat till ett läkemedel som blockerar SSL5-proteinet som hindrar immunförsvaret från att angripa bakterien Staphylococcus aureus.
Tillvägagångssätt 2
- Leta fram antikroppsfragment som binder vid SSL5 från antikroppsbiblioteket.
- Framställ fragmenten som binder.
- Kolla om de återhämtar MMP8:s funktion genom att hämma SSL5.
- Om de fungerar har du kringgått hela den strukturbaserade designprocessen och kan genast börja fundera på läkemedel.
En tvådimensionell bild av en tredimensionell modell
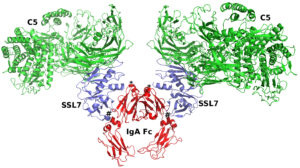
Bilden visar ett komplex av flera proteinstrukturer. Bilden kommer från en tredimensionell digital modell som går att vrida och ändra, förstora och så vidare.
Det blåa proteinet är SSL7 – Staphylococcal Superantigen-Like protein 7 som är en virulensfaktor av bakterien Staphylococcus aureus. SSL7 är lik, men inte identisk med SSL5 som diskuteras i texten.
SSL7 binds till åtminstone två olika proteiner i människans immunsystem för att störa normala immunförsvaret:
- Det ena proteinet är komplementkomponent C5 (grön) som vanligen deltar i immunförsvarets aktiveringskaskad. Komplement C5 är en del av en process som gör så att ett hål borras till bakteriecellmembranet och bakteriecellen lyseras. Att bakteriecellmembranet lyseras betyder att det upplöses, bakterien dör.
- Det andra proteinet är immunoglobulin A (IgA, röd) som spelar en viktig roll i det mänskliga slemhinneförsvaret (det mukosala immunförsvaret).
Man har kristalliserat båda bindningskomplexen: SSL7 med IgA och SSL7 med C5. Strukturerna kommer från Protein Data Bank och har PDB ID-koderna 2QEJ och 3KLS – dessa finns på webben för den som är intresserad.
På bilden ser vi en modell där två SSL7-proteiner samtidigt binds till två C5 och ett IgA. Modellen baserar sig på experimentella data som visar att det är möjligt att ett sådant komplex kan existera.
En av de viktiga sakerna man får ut av bilden: man kan se att SSL7 binds med de här två proteinerna och när det binds till dem blockerar det också bindningen av två immunsystemets proteiner till deras naturliga partners IgA och C5. C5 borde bindas till stället som är märkt med * och IgA Fc-receptor (FcαRI) borde bindas till stället som är märkt med #. Att de inte gör det, att SSL7 kommit emellan, stör det normala immunförsvaret.
Protein Data Bank (PDB)
Protein Data Bank är en världsledande digital open access-resurs för forskning inom biologi och medicin. PDB nås genom internet och ger tillgång till tredimensionella strukturer på stora biologiska molekyler (proteiner, DNA och RNA).
Superdatorn Puhti
CSC, IT Center for Science, är ett ickevinstdrivande bolag som bistår forskningen i Finland med internationell standard på högkapacitetsberäkningar. Den nyaste superdatorn heter Puhti, ett BullSequana X400-system levererat av teknologiutvecklingsbolaget Atos. Systemet stöder en bred repertoar olika typer av beräkningsuppgifter, som simulationer, högkapacitetsberäkningar, data-analys och djupinlärning.
Puhti består 682 CPU-noder med en minneskapacitet på 192 gigabyte till 1,5 terabyte. Varje nod har två Intel Xeon Gold 6230 Cascade Gold-processorer och varje processor har 20 stycken kärnor på 2,1 gigahertz. Puhti har en teoretisk effekt på 1,8 Petaflops och en minneskapacitet på 4,8 petabyte (4,8 miljoner gigabyte).
I Alankos och Salo-Ahens forskning behövs Puhti bland annat för att behandla de enorma mängder data som behövs för att sålla fram och analysera lämpliga molekylstrukturer.